Vector_NTI_中文使用说明书

Vector NTI 7.0 User's Manual
软件包中文翻译者:宋厚辉(浙江大学)
前言(Introduction)
1.程序附带的数据库(V ector NTI database)包括:DNA/RNA、蛋白质、内切酶、寡核苷
酸、凝胶mark。此外程序还提供数据库开发(Database Explorer)功能,用户可以自己修改、添加、拷贝感兴趣的各类数据库。
2.创建新分子(有四种方法)
A.用GenBank/GenPept, EMBL/SWISS-PROT and FASTA、ASCII等格式输入DNA 或氨基酸。
B.手工粘帖,然后保存到数据库中
C.从其他分子、接头、载体中剪切、拼接构键
D.从DNA或RNA分子的编码区翻译成蛋白质
3.关于新分子的序列特征图谱:利用GenBank/GenPept, EMBL/SWISS-PROT or FASTA等
格式输入的分子都能显示出序列和结构图,但自己手工粘帖的没有,需要自己编辑
第一章Chapter 1
Tutorial: Display Windows(显示窗口)
目的:创建显示窗口,并对图、序列和文本进行操作
? 1.登录Vector NTI
安装后首次登录,系统将提示是否允许填充空库,点OK。这样DNA molecules, proteins, enzymes, oligos, and gel markers将组成NTI的数据库。并出现下列两个窗口。
? 2. 观察出现的Vector NTI 工作窗口和Database Explorer窗口
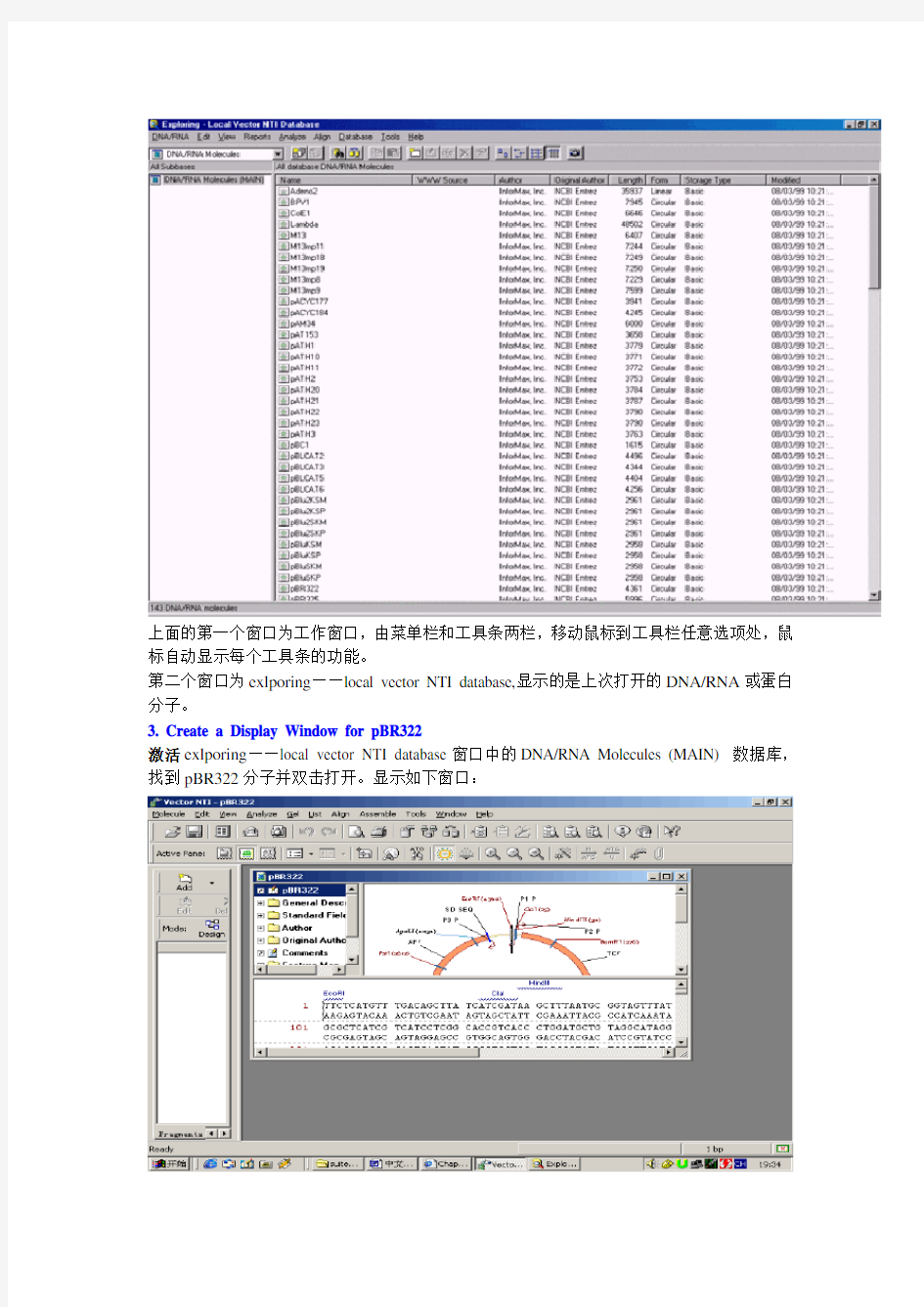
上面的第一个窗口为工作窗口,由菜单栏和工具条两栏,移动鼠标到工具栏任意选项处,鼠标自动显示每个工具条的功能。
第二个窗口为exlporing——local vector NTI database,显示的是上次打开的DNA/RNA或蛋白分子。
3. Create a Display Window for pBR322
激活exlporing——local vector NTI database窗口中的DNA/RNA Molecules (MAIN) 数据库,找到pBR322分子并双击打开。显示如下窗口:
? 4. 观察pBR322 显示窗口
上面的窗口由文本区(对分子信息的文字描述,双击文件夹可以看到)、图形区(标住限制性内切酶位点等)和序列区(全序列及酶切位点)三个部分组成。
? 5. 显示窗口的管理(通过拖拉标尺,改变窗口、或每个显示区的相对大小)? 6. 转换pBR322’s 图形区:在工具栏左边的active pane右侧有三个按钮,用鼠标点当中那个(graphics pane)
?7. 对pBR322’s 结构图进行操作:用户可以尝试点击、、,此时图形的大小会发生变化。选区设定:菜单Edit——>set selectio n,输
入100bp–1000bp,然后OK.可以看到选取在图形中用扇型框圈了起来.将
鼠标移到选取的5’端,可以看到,通过拖拉可以延长或缩短选区范围,同样
在3’端也能做到。如果用户一次只想移动一个碱基用直接拖拉就可能不方
便,假如用户想在5’端移动一个碱基,首先将鼠标放到处,然后按住shift
和键盘右侧的——>或<——箭头,则按一次箭头移动一个碱基距离。如果
一次想移动10个碱基,则同时按住shift+ctrl+箭头。如果用户只是粗略选
择,可以直接将鼠标移到图中,用十字花拖动。将鼠标移到图的TCr处,此
时鼠标箭头变成,并显示TCr代表的含义,如果用户此时按下鼠标,则
TCr的编码区将被选中。
?8. 检查pBR322’s nucleotide 序列
移动标尺,尽可能将更多的PBR322序列显示出来,并点击序列中任何一处。
现在对序列显示的样式进行设定:点击(Display Setup)按钮,显示molecular display setup对话框:
用户可以对序列的颜色、大小、10个一组显示还是15个一组(默认是10个碱基一组),结构图的颜色、限制酶切图谱(注意刚开始显示的PBR322上限制酶并不多)、ORF、Motif等进行设置。现在我们打算把序列字体的颜色由黑色变成绿色,显示全部PBR322的酶切图。操作如下:在刚才的窗口中点restriction map下面的RMap setup按钮,在出现的对话框中点Add,再在出现的对话框中点select all,然后OK。点sequence下面的sequence setup,可以看到序列长度的设置等,在color栏中选green,一路点OK。此时显示如下:
我们发现限制酶是大大的多了,不过美中不足的是序列显示的是两条链(正链和互补链),实际上一条链就够了,还有最好再和编码的氨基酸一切显示。这好办:
先选中全序列(Ctrl-A),看到工具条中的(Translate Direct)图标
了没,点它。哈哈果然翻译成功了。不要忘了看左右两侧的图标哟,点点看。(注意用户在发表文章的时候,一般在文章中发表自己克隆或表达的DNA 序列,在DNA序列下面还有氨基酸序列,哈哈,这不帮你做到了。什么?内切酶怎么去掉,刚才你怎么加上去的就怎么解除吧,看看我下面的图不就是办到了):
9. 对pBR322’s text 文本描述进行操作
拖动标尺,使文本区尽可能拉大。选中Restriction Map文件夹,然后找到工具条中的Expand Branch按钮,点它。其实这和双击restriction map
文件夹是一样的,都是打开的意思,还有它左边的按钮。在找到Feature Map
文件夹,然后按按钮。看到TC(R)了没,记住它。这是一个四环素抗性基因,在第7章中将详细叙述如何将这段基因克隆到载体PUC19中。
10. 将pBR322’s 文本区和图区、序列区连接起来(可以让你一个一个的细细品位PBR322的每个细小结构,全部看可能会眼花,那就一个一个的看吧)
激活文本区,然后找到(Link Panes)按钮,点它。完了,PBR322的图区上的任何标记都没了,成了一个圆圈。还有,序列区的酶切标记也没了。哈哈,
不用急,先点文本区的Restriction Map文件夹,然后点按钮打开文件
(Standard Arrangement)按钮了没,点它。会发现酶切图谱显示的方式
和刚才不一样了,这是标准方式。在文本区中选中feature map文件夹,点
打开,发现图形又变了。依次关掉feature map中的其他文件夹,只留
TCR,此时图中只有TCR一个标记了。最后别忘了再点一下链条,发现图又回到原样。如下图:
(看看上面的时钟都23:12了,该睡觉了),明天继续。
11. 打印pBR322’s 文本description, 图形map, 和序列sequence
打印文本:先激活文本区,(就是active pane右边的第一个按钮,或者直接用鼠标在本文区点一下),然后按expand branch按钮打开文本区内的所有文
件夹,然后点打印。同样要想打印图形或序列,先激活其所在的选区,然后按打印机图标即可。
12.为41BB_HUMAN创建显示窗口
点击窗口下面的exploring——local vector NTI database图标,打开打开Explorer窗口,点击窗口左上角的下拉式菜单,选Protein Molecules (MAIN)数据库。找到41BB_HUMAN’s并双击。打开窗口如下:
窗口显示结构和DNA序列的显示窗口一致,也包括文本区,图形区和序列区三部分。菜单和工具条也基本一样。在文本区中双击Analysis文件夹,则蛋白自动分析结果以表格的形式在下面显示出来。下面我们把这两个表格拷贝到word文档中,先用shift+鼠标将两个表格选中,然后点工具条中的照相机(camera)命令,在出现的对话框中可以看到序列的range中的selection已被选中,点Copy.,然后打开一个word文档,粘帖(ctrl-V),则表格被完整的拷贝到word文档中了。如下所示:
Length 255 aa Molecular Weight 27897.66 m.w.
1 microgram = 35.845 pMoles
Molar Extinction coefficient 11250
1 A[280] corr. to 2.48 mg/ml
A[280] of 1 mg/ml 0.40 AU Isoelectric Point 8.13 Charge at pH 7 3.72
Amino Acid(s) Number count % by weight % by frequency
83 36.68 32.55 Charged
(RKHYCDE)
Acidic (DE) 25 10.85 9.80 Basic (KR) 29 14.43 11.37 Polar (NCQSTY) 90 34.08 35.29
67 27.06 26.27 Hydrophobic
(AILFWV)
A Ala 11 3.02 4.31
C Cys 25 9.33 9.80
D Asp 11 4.51 4.31
E Glu 14 6.34 5.49
F Phe 16 8.14 6.27
G Gly 21 4.85 8.24
H His 2 0.96 0.78
I Ile 7 2.83 2.75 K Lys 13 5.85 5.10 L Leu 21 8.48 8.24 M Met 3 1.38 1.18 N Asn 12 4.88 4.71 P Pro 18 6.38 7.06 Q Gln 12 5.40 4.71 R Arg 16 8.58 6.27 S Ser 22 7.12 8.63 T Thr 17 6.24 6.67 V Val 11 3.97 4.31 W Trp 1 0.63 0.39 Y Tyr 2 1.12 0.78 B Asx 23 9.39 9.02 Z Glx 26 11.74 10.20 X Xxx 0 0.00 0.00
13. 为1B14_HUMAN创建显示窗口
重新回到exploring——local vector NTI database窗口,找到
1B14_HUMAN分子并双击打开。如下图:
注意该蛋白分子图形上的各种特征显示的十分紧凑,大有眼花缭乱的感觉,为了方便起见,我们可以按刚才介绍的link命令来逐一显示各个feature。操作方法和DNA分子一致。
关闭窗口,结束,当最后一个窗口关闭是屏幕提示this will end your vector NTI session,点确定(OK)关闭。
第二章:Chapter 2
Tutorial: Molecule Operations分子操作
目的:对pBR322 的general data, feature map, and sequence进行编辑(注意蛋白质和DNA分子的操作是一样的)
1.登录Vector NTI程序(刚刚说完,不用再教了吧)
2.打开pBR322的显示窗口(再罗嗦一遍吧,程序vector NTI——
Exploring local vector NTI Database——DNA/RNA
Molecules (MAIN)——PBR322,双击。)
3. 对pBR322’s 的常用数据(general data)进行编辑
在文本区的最上面,双击PBR322名字,弹出下面窗口:
1st:给PBR322加关键词:点keywords,在弹出的关键词窗口中输入My own plasmid,点Add。回到DNA/RNA Molecular 窗口,将最下面的description中的内容替换成My pBR322。点OK(确定)。注意屏幕的左上角pBR322*,在PBR322的后面有一个星号,说明现在显示的是PBR322的修饰形式。现在我们要在数据库中保存这一结果:
菜单molecular——save as,在弹出的对话框中输入序列的名字My
pBR322,点OK。这时发现星号不见了,说明结果已保存到数据库中,这时数据库中关于PBR322的DNA分子有两个,一个是原始的PBR322(就是最初打开的那个),另一个就是我们保存的那个my PBR322。
3.编辑My pBR322’s序列
激活序列区,菜单edit——set selection,在对话框中输入范围21-40,点OK。会发现序列选区内含有ClaI 和HindIII两个酶切位点。点击菜单edit——
new——Replace Sequence 21 bp–40 bp,将窗口中第23和24位的TC删除分别用AA代替,窗口将显示如下:
注意该窗口左下脚显示有:inserted 2,delete 2,什么意思都不用说了。点OK,注意序列中的ClaI位点立刻消失了。如下图所示:
5. 将My pBR322的修改结果取消,不保存到数据库
注意刚才修改完后,在屏幕左上角My pBR322的后面,有一个星号,说明当前显示的分子已经修改,下面将修改结果取消,点击菜单mo lecular——Revert To Saved,点OK确定。此时数据库将刚才的修改结果取消了。(如果需要保存修改结果则在molecular菜单中选save as命令)
6. 如何插入新的序列片段
通常在编辑序列的时候需要在序列图谱中插入一段基因或者一段特征序列,先找到序列中的AP(R) 标志(3293 bp–4156 bp)和TC(R) 标志(86 –1276 bp),菜单edit——Set Caret Position,输入200,将光标打到200bp处,下面我们要在此位置处输入10个T碱基。点菜单edit——new——insert sequence 200bp,在出现的对话框中输入10个T,点OK,然后又出现一个对话框,问你是否确认当前的序列已经修改(CDS TC(R) is affected by sequence editing,Delete, Delete All, Keep, and Keep All),点keep.我们发现在200bp序列处多了10个T。我们将鼠标移到AP(R)处,我们发现其位置已经顺时针移了10个碱基(3303 bp–4166 bp)。其实,在原始序列中一旦插入一段序列后,系统会自动改变图谱中各特征的相对位置,如果插入的序列位于某一特征序列的内部,我们点Keep,NTI 会自动向3’端移动。现在我们将鼠标移到TCR处,发现其3’端已经后移了10个碱基(1286),不过5’端没动哟。如下图所示:
7. 编辑TC(R) 信号特征
将鼠标移到图形的TCr 处,当箭头变成“手”的形状时双击(或者点鼠标右键,选feature properties),在出现的对话框中用户可以修改TCr的位置、名称和描述。我们将名称(name)由TC(R)改成Old TC(R),然后在最下面的
description中输入描述:“10 bp fragment inserted”,点OK。则图形结构变成:
8. 删除P2_P信号,并加入新的序列特征
在图谱中找到P2P,选中,点鼠标右键,选Delete Feature From Fmap (或者从edit菜单中选择此命令),此时NTI将提示P2_P will be deleted from the feature map,点OK(确定)。我们发现图谱中P2P已经没有了。
下面我们我为PBR322序列加入一个新的特征:
edit——set selection——输入3000-3500bp,点OK。在工具条中找到
(add features)按钮,点它。(也可以从edit——new——add feature to FMap进入)。在出现的对话框feature name中输入New Feature,注意NTI默认的特征类型(feature type)为Misc. Feature,用户可以从列表中
选择自己认可的特征。最后点OK。如图:
下面将My pBR322的修改结果保存到数据库中:菜单molecular——save as,(如果不想改名的话)点OK,NTI将提示My pBR322已经存在,是否覆盖,点overwrite.
9. 修改My pBR322的起始坐标(这样可使刚才插入的10bp片段和最初的PBR322具有一致的坐标系)
菜单Molecule——operations——Advanced (DNA/RNA)—— Change Starting Coordinate。在出现的对话框new start(新的起始点位置)输入:11(因为刚才插入了10bp,所以起始点是1+10=11)。点OK,此时NTI会提示当前坐标已被修改,是否继续,点OK确定。现在我们会发现显示窗口中TCR 的位置已经变成了(76bp–1276bp),还有AP(R)的位置(3293 bp–4156 bp),这都和最初的PBR322一致。如下图:
10.退出显示窗口,关闭NTI
第三章:Chapter 3
Tutorial: Working With A Molecule’s Graphical Representation(对分子图形的
展示进行操作)
目的:以DNA分子为例,对分子图形的展示进行操作(蛋白质的操作和DNA 类似),为pBR322图形创建一个展示窗口,并保存到分子文档中
1.登录Vector NTI程序
2.通过新窗口打开PBR322(与前两章的打开方式略有不同)
在NTI主窗口中,点击工具条最左边的打开文件夹(或者mo lecular——Open),在弹出的Open窗口中点击database DNA/RNAs,在列表中找到PBR322,然后OK。
3.显示窗口的调整
比如我们想观察图象的详细情况,首先用标尺拖动图形窗口,至于序列和文本区,
可以很小,因为我们的目的是看图。然后点或让图形放大或缩小,直到
满意为止,如果用户想一步步的看放大效果,可以按住shift的同时点。
4.修改图形的自动排列设置
按住ctrl的同时,点(Standard Arrangement),用户可以根据弹出的小菜单,来选择图形线条的粗细和字体的大小,直到满意为止。
5.修改图形的编码区信号(CDS signals)设置
点中图形中任意编码区(粗箭头),然后按鼠标右键,在弹出的下拉菜单中,选CDS Display Setup,用户可以在弹出的Graphics Display Setup对话框中修改所选编码区的名称、颜色标记、箭头的粗细等,(实际上NTI默认的就很好了)。用户也可以通过点more来进行更多的修改(比如字体和大小等,和word中字体
的处理很相似),修改完后一路OK点下去即可。注意这种修改只是针对本窗口中的分子,对其他分子没有影响。比如我们将箭头填充成兰色。下面我们保存这一设
置:点击(display setup)右侧的小三角形符号,在弹出的菜单中选Save Settings As,然后在弹出的窗口中给刚才的设置风格取个名字,不妨叫blue,
点OK,注意此时NTI 会提示是否保存其他没用过的风格,点NO。现在再点击(display setup)右侧的小三角形符号我们会发现blue已经在下拉菜单上了。(注意这种改变一改就是好几个箭头一块改,如果只想改一个箭头的颜色,请看第6条)
6.打开图片编辑方式
NTI提供了两种编辑图片的方式,分子编辑方式(默认)和图片编辑方式。激活图
形区然后按(edit picture)按钮。然后点中图形中任意标记或者箭头,按住鼠标右键,在弹出的下拉菜单中选properties(属性)或者style(风格)等,进行设置,注意此时设置的仅仅是所选的箭头或者标记,而不是整个分子(在第5条中设的确是整个分子,请注意比较,也就是第5条的方法是分子编辑方式,两种方式
的转换通过按钮)。
7. 将TC(R)箭头变成兰色网格
操作如下:点中按钮,点中TCR箭头,按住鼠标右键,选properties——
fill,按右图方式选择:
点OK(如果是中文操作系统,OK=确定)
8. 放大TC(R)箭头
点中TCR后,当鼠标在箭头附近移动时,会出现两种十字形标记:和,通
过鼠标拖动可以改变箭头的胖瘦,而拖动则可以改变箭头的相对位置(移到
圆内或圆外)。如果想让箭头按圆圈转动,可以在按住ctrl+shift同时,拖动。
注意这种拖动并不能修改分子本身,仅仅是改变位置而已,如果想修改分子中的标记,请使用第二章介绍的方法。现在,拖来拖去是不是将箭头拖的乱七八糟了,那
就按取消吧。如下图:
9. 修改TC(R)标记的格式
将鼠标移到TCr标记上,双击。显示下面的对话框(就是属性)
点Font,选择斜体18号字,点OK。TCR将显示如下图:
我们发现,字体大了。点(Standard Arrangement)按钮,使标记的信号回到标准位置,如果图片已经修改,NTI 还会不厌其烦的提示是否继续,点确定吧。
10. 加入文本注释
看到窗口工具条最右侧的“回形针”符号了没,点它(说什么,没反应?哈哈,那
你肯定把忘了,如果没反应,点后再点回形针肯定行),在弹出的对话框中输入“Clone into pUC19”,点OK。此时加入的注释出现在圆圈的中央,用鼠标拖到TCr的下面即可(什么?字体太小,哈哈,点中鼠标右键从properties 中选择字体的大小和颜色)。如下图:
如果觉得不爽,可以先选中刚才添加的文本,点菜单edit——Delete Annotation 进行删除。注意以上修改的仅仅是显示的界面,NTI并不能将修改的显示结果保存到数据库中,所以用户也可以发现在左上角并没有*号标记,也就是数据库中的分子没有改变,如果想让它改变的话,只能将文件存到文档文件中了。
11. 将pBR322 分子显示结果保存到文档文件中
菜单Molecule——save as——save as file,名称NTI已经给起好了,就是pBR322.gb。选择好要保存的目录,点OK。如果感觉刚才创建的blue比较爽,
可以在保存前选择右侧的三角按钮选择blue。此时系统会提示是否创建html文件描述分子,点yes(这样我们可以和朋友通过internet进行交流了)。注意该文档完全与数据库相关联,其显示的信息与数据库中的信息一致。但风格与数据库不同,比如箭头的颜色、字体的大小等(当然由用户决定)。现在用户可以先关闭文档然后再打开就可以发现显示的风格和数据库中的风格完全不同(不过内容还是一样的)。注意:对于原先数据库中没有的分子(比如用户根据自己的需要自己构建或从GENBANK下载的,并不能直接修改显示风格,如果想修改其显示风格,先将该分子保存到数据库中,这样就可以了)
退出程序
第四章Chapter 4
Vector NTI 工具和internet连接
目的:将本地数据发送到internet上,并进行BLAST 搜索, 序列对排、比较和分析等
1. 登录Vector NTI
2.在新窗口打开pBR322
3. 通过exploring local vector NTI database窗口(切记)选中pBR322整个分子并利用BLAST 搜索工具进行序列比较(注意:如果不是通过exploring 窗口,而是通过NTI主窗口“打开”命令,打开PBR322,则最终的搜索结果不会直接显示在屏幕上,而是发到用户指定的email中,但结果都是一样的)
选中后,点exploring local vector NTI database窗口中的Tools——Compare Against——GenBank via BLAST on NCBI Server,在弹出的菜单中按下面窗口选择全序列和正链并点OK确认(注意保证网络连接正常,否则无法连接到Genbank):
当连接成功后,将弹出NCBI入口口,如下图所示(不过由于使用的internet浏览器不同,可能每个人显示的窗口可能不一样,我使用的示IE5.0):
点blast,弹出如下窗口:
点format,此时NCBI将正式开始搜索(注意这个过程可能需要几分钟,请耐心等待,窗口下面会显示该过程需要的时间,比如This page will be
DNAStar详细中文使用说明书
Sequence Analysis Software for Macintosh and Windows GETTING STARTED Introductory Tour of the LASERGENE System MAY 2001
DNASTAR, Inc. 1228 South Park Street Madison, Wisconsin 53715 (608) 258-7420 Copyright . 2001 by DNASTAR, Inc. All rights reserved. Reproduction, adaptation, or translation without prior written permission is prohibited,except as allowed under the copyright laws or with the permission of DNASTAR, Inc. Sixth Edition, May 2001 Printed in Madison, Wisconsin, USA Trademark Information DNASTAR, Lasergene, Lasergene99, SeqEasy, SeqMan, SeqMan II, EditSeq, MegAlign, GeneMan, Protean,MapDraw, PrimerSelect, GeneQuest, GeneFont , and the Method Curtain are trademarks or registered trademarks of DNASTAR, Inc. Macintosh is a trademark of Apple Computers, Inc. Windows is a trademark of Microsoft Corp. ABI Prism are registered trademarks of Pharmacopeia, Inc. Disclaimer & Liability DNASTAR, Inc. makes no warranties, expressed or implied, including without limitation the implied warranties of merchantability and fitness for a particular purpose, regarding the software. DNASTAR does not warrant, guaranty, or make any representation regarding the use or the results of the use of the software in terms of correctness, accuracy, reliability, currentness, or otherwise. The entire risk as to the results and performance of the software is assumed by you. The exclusion of implied warranties is not permitted by some states. The above exclusion may not apply to you. In no event will DNASTAR, Inc. and their directors, officers, employees, or agents (collectively DNASTAR) be liable to you for any consequential, incidental or indirect damages (including damages for loss of business profits, business interruption, loss of business information and the like) arising out of the use of, or the inability to use the software even if DNASTAR Inc. has been advised of the possibility of such damages. Because some states do not allow the exclusion or limitation of liability for consequential or incidental damages, the above limitations may not apply to you. DNASTAR, Inc. reserves the right to revise this publication and to make changes to it from time to time without obligation of DNASTAR, Inc. to notify any person or organization of such revision or changes. The screen and other illustrations in this publication are meant to be representative of those that appear on your monitor or printer.
DMG 海德汉iTNC 操作培训
DMG培训照片文件整理总结(详见机床操作说明书) 目录: 一.手动拆刀和装刀: 1.进入手动模式。 2.按刀具表软件,进入刀具表。 3.打开编辑开关。 4.建刀库刀位以外的刀具。 5.按结束。 6.进入MDI模式。 7.调用刚建立的刀具。 8.按START键执行。 9.显示更换刀具。 10.按开门键,打开工件间门。 11.按换刀键。 12.屏幕T 开始闪烁。 13.旋转按刀按扭,听到有松夹声音。 14.注意刀具的缺口方向,放刀具到位,松开换刀按扭,听到夹紧的声音,松开刀具。 15.刀具在主轴。 16.关闭工作间的门。 17.屏幕显示更换刀具。 18.刀具已经换入。 19.确认换刀完成。 20.换刀结束, 二.手动拆除刀库以外的刀具。 1.按MDI 进入 2.调用零号刀具。 3.按START 键执行。 4.屏幕显示从轴上取下刀具。 5.按开门键开工作间门。 6.按换刀键。 7.屏幕T 开始闪烁。 8.用手拿住刀具。 9.旋转拆刀旋扭,拆除刀具。 10.关闭工作间的门。 11.屏幕显示 T0 。 12.屏幕显示从轴人取下刀具,按START 键完成拆刀。 三.从刀库装入刀具。 1.按手动按扭,进入手动数据输入。 2.按刀具表软键,进入刀具表。 3.把光标移到要装入刀具的一行。 4.按左边的刀库管理软键。 5.按刀具拆除, 6.等待屏幕显示 1。20(1号刀库,20号刀位),刀库已经准备好。 7.因为只是装刀,并不是真正拆除,所以按中断结束。 8.打开后面的刀库门,放入刀具,注意缺口方向在里面,完成刀库装刀。
2.把光标移到要拆除的刀具的一行。 3.按刀具管理软键。 4.按刀具拆除。 5.屏幕显示(1。32)一号刀库32号刀位。 6.按中断软键,不清除刀具参数。 7.打开刀库门,拆除刀具。 8.手动清除刀具数据,按结束,完成刀库拆除刀具。 五.标准刀的校正Z零点。 1.把标准刀放入32号刀位。按MDI 进入。 2.调用32号刀具。 3.按START 执行。 4.调入标准刀具。 5.在刀具表中输入,标准刀的长度和半径。 6.移动标准刀在工作台上方50MM 处。用50MM的标准块测量,精度到0。001MM。 7.进入设定原点界面。 8.按设定原点软键。 9.输入Z+50 10.按ENT 键确认。 11.按预设表软键,进入。 11.进入预设表 12.把工作台设为Z零点。标准刀的校正Z零点完成 (工作台有-0。0397的误差,会在后面,五轴精度校准中校正) 探头校正方法 1. 用基准刀找到机床工作台面的机械坐标.(用50mm标准块) 2. 调出探头,把探头移动到距离工作台面20MM的地方,执行探头长度标定(该选项在打开探头功能看到两个标定的第一项)。标定完成后会看到探头的实际自动的写到刀具补偿里面。 3. 用机床配置的内径为50的环规做探头摆动的标定。做法为把探针动到环规的上表面-10MM左右的地方执行探头半径标定,{该选项在打开探头功能看到两个标定的第一项}(做这项这前要用千分表测出探头放在主轴上的摆动值不能超过5μm如果摆动太大要用装探头的木盒配的六角匙调整探头下表面的四个螺母,调整后探头的摆动越少越好) 六.探头直径校正 1.调用探头,用丝表校探头最高点在0。005MM以内,在刀具表中建立探头数据,L (探头长度)R(探头半径)和PLC参数00010100。 2.如果探头不正,用六角松开校正。 2.用铜棒或木棒轻轻敲击校正。 4,校正好后,按探测功能软键。 5.选择标定R功能。 6.把探头移到环规的中心。 7.记录环规的直径。 8.输入环规的半径和探头的半径。 9.按+X 软键,确定方向。 10.按START 执行测量。 11.屏幕显示X+的测量数据。 12.按START 键执行下面的测量。 13.按180 度软键, 14.START 执行 15.完成测量
DM中文使用说明书
DMX512中文使用说明书 (2009-03-20 16:07:26) 转载 分类:灯光设备使用和技巧 标签: 灯光设备 一、四位数码管说明: XXXX *第一位代表CHASE,共有6个 *第二位代表SCENSE,共有8个 *第三四位代表BANK,共有30个 *设置MIDI通道时第三四位代表MIDI通道,共有16个MIDI通道 *一个CHASE最多可以包含240个SCENSE *一个BANK最多可以包含8个SCENSE *一个SCENSE最多可以包含192个通道(也可以说是12个SCANNER) SCANNER1:通道1~通道16 SCANNER2:通道17~通道32 以此类推 SCANNER12:通道181~通道192 *一个SCANNER最多可以包含16个通道 *调节滑杆时显示数值或者百分比 二、操作时请注意数码屏的指示灯在什么状态。 *BLANKOUT *STEP *PROGRAM *MUSIC TIGGER *AUTO TIGGER 三、DMX512面板功能说明 1、SCANNERS 按下SCANNER键,其旁边的LED灯亮,其中连接8个通道的输出可被调节,在SCENS运行时,如果可调电位器控制为OFF,则调节电位器不会影响通道输出,但如果可调电位器控制为ON,则通道输出会随相应的可调电位器的改变而改变; 2、SCENS按健 按下一个SCENS键可触发SCENS或存入一个SCENS,第二个数码管头显示SCENS1-8; 3、可调电位器 调节可调电位器改变DMX的通道输出大小,最小是0最大为255或者从0%-100%,可调电位器1-8控制连续的八个通道;
4、PAGE/SELECT键 选择PAGE A或PAGE B,PAGE A为每个SCANNER的前八个通道PAGE B为每个SCANNER的后八个通道 5、SPEED SLIDER 推动这个推杆调整走灯速度; 6、FADE TIME SLIDER 推动这个推杆调整FADE TIME; 7、LED DISPLAY 8、BANK按键(↑/↓) 第三位和第四位数码管显示BANKS(01-30),按下↑/↓键,BANK增大或减小,显示的SCENS为该BANK里的SCENS; 9、CHASE 1-CHASE 6键 用于CHASES编程或CHASES运行的选择; 10、PROGRAM键 上电本机在走动运行状态,按下PROGRAM键盘2秒,编程指示灯闪动可编程SCENSR和CHASER,再按下PROGRAM键2秒,编程指示灯灭回到运行状态; 11、MIDI/ADD键 A、在运行状态按住MIDI键2秒,第3及第4位数码管闪动,通过↑或↓选择MIDI通道,再按MIDI键2秒结束MIDI通道的设置选择的MIDI通道被存贮;或者除↑/↓键以外的任何键都可结束MIDI通道的设置,不存贮所选取的MIDI通道; B、在编程状态,用于编辑; 12、AUTO/DEL键 A、在运行状态,按下AUTO/DEL键,自动触发指示灯亮,表示在自动触发状态,再按下AUTO键退出自动触发状态,自动触发指示灯灭; B、在编程状态,用于SCENS及CHASE编程; 13、MUSIC/BANK COPY键 A、在运行状态,按下MUSIC键,声音触发指示灯亮,可由声音触发SCENS,再按一下MUSIC键,声音触发指示灯灭,退出声音触发状态; B、在编程状态,用于SCENS及CHASE编程;
RTKLIB中文说明书
1.文件目录结构 \app-- APs构建环境 \bin --可执行二进制APs和windows链接库 \data-- APs样本数据 \doc --文档文件 \lib --库生成环境 \src --RTKLIB库的源程序 \test--测试程序和数据 \util-- 实用程序工具 2.\bin\rtklaunch.exe 应用程序启动器 3.RTKNAVI实时定位结算 输入GPS / GNSS接收机原始观测数据,实时进行导航处理。 3.1执行\bin\rtknavi.exe
3.2用RTKNAVI进行实时定位必须输入GPS/GNSS接收机原始观测数据和卫星星历,点击I进入输入流对话框 检查设置Rover、Basestation、Correction三个选项的设置,如果设 置定位模式,只选择一个,基站和校正并不需要。 流类型可有从以下选项中选择 (a)Serial :串口输入数据 (b)TCP Client :连接到一个TCP服务器,通过TCP连接输入数 据 (c)TCP Server :接受一个TCP客户端连接和通过TCP连接的输 入数据 (d)NTRIP Client :连接一个NTRIP caster输入数据 (e)File :日志文件中输入数据。[.conf] (f)FTP :通过FTP下载一个文件后输入数据 (g)HTTP :通过(a) HTTP 下载一个文件后输入数据 3.3选择流类型为?Serial?(连续的)点击...按钮设置选项
3.4在流类型中如果你选择了SerialTCP Client或者TCP Server作为类型,你可以通过流设置GPS / GNSS接收机启动和关闭命令,设置命令,按下“Cmd?标签下的…按钮。在?Serial/TCP Commands?对话框中进行设置,可以加载和保存命令 3.5流类型中设置类型为?File?可以设置文件输入路径,数据为原始数据,还可以设置时间 3.6设置输出流格式,点击O按钮,弹出 ?Output Streams?对话框,设置类型,
1海德汉中文使用说明书[1]
1前言
1.1TNC 426,TNC 430 HEIDENHAIN TNC是一种面向生产车间的仿型控制器,使您能以一种便于使用的对话式编程语言,编制使机床准确加工运转的对话式程序。TNC控制器可用于铣削、钻孔和镗削加工,也可用于加工中心。TNC 426最多可控制五根轴;TNC 430最多可控制九根轴。您也可在程序控制下改变主轴的角度位置。 一体化的硬盘能存储许多您所喜欢的程序,不论这些程序是脱机创建的还是数字化的。为了能快速计算,随时随地都能在屏幕上调出袖珍计算器。 键盘和屏幕布局清晰合理,功能调用快捷,使用方便。 编程:HEIDENHAIN对话式和ISO格式 HEIDENHAIN对话式编程是一种特别容易的程序写入方法,交互式的图形表示仿型编程的各个加工步骤。如果某一张生产图纸没有标注NC适用的尺寸,HEIDENHAIN FK任意形状轮廓编程就会自动执行必要的计算。工件的加工状况,无论是现在正在加工中还是在加工之前,都能用图形模拟显示。在ISO编程格式或DNC模式中都由此功能。 当TNC在运行另一段程序时,您也可输入或测试一段程序。 兼容性 TNC能执行所有写在TNC 150B及以后的HEIDENHAIN 控制器上的零件程序。
1.2可视显示器和键盘 可视显示器 TNC显示器可使用CRT彩色显示器(BC120)或TFT 液晶显示器(BF120)。右上图为BC120的键盘和控制器,右中图为BF120的键盘和控制器。 屏幕端部 当TNC接通电源时,屏幕端部显示选定的操作方式:左侧为加工方式,右侧为编程模式。当前激活的模式显示在一个较大的方框中,在此方框中,同时也显示对话提示和TNC信息(如果没有,则仅显示图形)。 软键 TNC底部一排软键表示辅助功能。直接按下这些键,即可选用这些辅助功能。紧接着软键行上面的行表示软键的编号,可以左右移动黑色光标调用。 被激话的软键行高亮显示。 软键选择键 切换软键行 设置屏幕布局 用于转换加工和编程模式的移位键 仅在BC120上的键 屏幕退磁:为屏幕设置退出主菜单 为屏幕设置选择主菜单: 在主菜单中:高亮显示部向下移动 在子菜单中:减小数值;图形向左或向下移动 在主菜单中:选择子菜单 在子菜单中:退出子菜单 主菜单对话功能 CONTRAST调节对比度 H-POSITION调节水平位置
绕膜机中文使用说明书
目录 一:拉伸薄膜缠绕包装机 (2) 二:主要技术参数 (2) 三:产品保修 (3) 四:拆箱、安装、调试 (3) 五:控制板操作说明 (5) 六:设备维护 (7) 七:使用 (9) 八:功能使用及其它 (9) 8-1、记数误差10 8-2、光电开关的使用10 8-3、薄膜操作简图11 8-4、预拉伸薄膜导出11 8-5、可移动限位块11 8-6、薄膜拉伸12 九:使用安全 (13) 附设备可能出现的故障及排除方法 电气原理图
一:拉伸薄膜缠绕包装机 1. 拉伸薄膜缠绕包装机是以LLDPE拉伸薄膜为包装材料,对多种货 物进行裹包的专业包装机器。 2. 使用我公司出品的系列薄膜缠绕拉伸包装机器,可以降低包装成 本,方便存储与运输,易于对包装材料(薄膜)进行回收,减少环境污染。这是一种现今较普遍流行的绿色运输包装方式之一。 3. 这种包装方式已经广泛应用于玻璃制造业、纸业、化工业、机械 制造业、食品业等各种不同行业,特别是在出口贸易货物运输中的集装箱已经得到广泛的应用。 4. 我公司出品全面的拉伸薄膜裹包机,除托盘基本型外,还出品圆 筒纸/帘子布型,线缆型,水平型、圆筒径向包装等多种机型。另外我们还出品牛皮纸、珍珠棉、普通PE膜等裹包机,如有需要请向我们的销售部门索取更详尽的说明和资料。 二:主要技术参数 1.包装规格: 型号最大货高(mm)最大托板尺寸(mm) TP1650F-L 2000 1200╳1200 2.承重:≤2000 kg 3.使用包装材料: 材料宽度厚度膜卷内芯孔膜卷外径LLDPE拉伸膜500 mm 17-35μm76.2mm(3英吋) ≤280 mm 4.工作电源:AC220V/50HZ 20A -1p。 务必使用单独固定电源,严禁使用临时线或与其他设备合用电源,电压不得低于200V,或高于250V,接线时请核对火线零线和地线(因电源不正确引起的电气损坏和其他损坏不在保修之
1226海德汉530系统编程和操作说明书
百度文库 - 让每个人平等地提升自 我 NC 软件 340 420-xx 用户手册 HEIDENHAIN 会话格式
可视显示器上的控制器 切换屏幕布局 在加工或编程模式之间切换 选择屏幕上功能的软键 切换软键行 输入字母和符号的打字键盘 文件名 注释 ISO 程序机床操作模式 手动操作 电子手轮 通过MDI进行定 位单步程序运行 连续程序运行 编程模式 编程和编辑 试运行 程序/文件管理器TNC功能 选择或删除程序或文件 外部数据传输在程序中 输入程序调用 MOD功能 显示NC错误信息的帮助文本 袖珍计算器 移动高亮区直接到程序块循环和参数功能 移动高亮区 直接到程序块循环和参数功能 进给速度/主轴速度倍率控制旋钮编程路径移动 切入/切出轮廓 FK自由轮廓编程 直线 圆的中心/极坐标极心 圆及圆心 圆及半径相切连 接的圆弧 倒角 圆角 刀具功能 输入和调用刀具长度和半径 循环子程序和程序段重复 定义和调用循环 输入和调用子程序和程序段重复标号程序中间程序停止在程 序中输入探头功能 坐标轴和编号输入和编辑 选择坐标轴或输入坐标轴到程 序中编号 小数点 改变算术符号 极坐标 增量尺寸 Q参数 捕捉实际位置 跳过对话问题删除字 确认输入并恢复对话 结束块 清除数字输入或清除TNC错误信息 中止对话删除程序段
百度文库 - 让每个人平等地提升自我 TNC 型号软件和特性 本手册说明了TNC按以下NC软件号提供的功能和特性 TNC型号NC软件号 iTNC 530 340 420-xx iTNC 530E 340 421-xx 后缀E表示TNC的出口版本TNC的出口版本具有以下限制 可同时在不超过4个轴上进行直线移动 机床制造商通过设置机床参数修改机床TNC可用特性本手册中描述的一些功能可能在您的机床上没有提供 您的机床上可能没有提供的TNC功能包括 3维探头探测功能 使用TT 130进行刀具测量 攻丝刚性 在中断后返回轮廓 请与您的机床制造商联系以熟悉您的机床的特性 许多机床制造商以及HEIDENHAIN提供TNC的编程课程我们推荐这些课程因为这是提高您的编程能力和与其他TNC用户共享信息和想法的有效途径 探头循环用户手册 在另外手册中描述了所有探头功能如果需 要该用户手册的拷贝请与HEIDENHAIN联 系手册ID编号369 280-xx 使用地点 TNC遵守EN55022规范对A类设备的限制并主要用于工业化区域
中文使用说明书
用户使用说明
目录 1.手机外观和按键说明2.使用手机存储卡做为U盘3.WLAN 4.蓝牙 5.电子邮件 GMAIL 电子邮件 6.拨号 7.信息 8. 通讯录 9. 浏览器 10.录音机 11.时钟 12.计算器 13.相机 相机 摄像机 14.图库 15.音乐 16.日历 17.收音机 18. 设置 19. 手机使用注意安全
1 .手机外观和按键说明 在任何的应用程序或界面上,按下此键可返回首页界面。 按下此键可开启动作清单,让您在目前的界面或选项菜单中执行动 作。 按下此键可返回前一个界面,或是关闭对话框、选项菜单、通知面板 或屏幕键盘。 按住此键可开启电话的选项菜单,然后您可以选择要锁定屏幕、关闭 手机,或将手机设成静音模式。 按此键可以增大音量。 按此键可以减小音量。 静音状态时按此键可以将手机调为振动状态。 进入相机界面,可切换至前摄像头自拍 2.使用手机存储卡做为U盘 若要从计算机传送音乐、相片和其它档案到您的储存卡,您必须先将手机储存卡设成U盘。
将手机储存卡设成U盘 1)选择“USB已连接”,可以装载U盘,可将音乐、相片和其它档案到您的储存卡或内置存储卡中。 2)选择“作为USB存储设备使用”可以打开右边的选项。 具体如下图所示: 3:有截图会显示的状态栏 3)插入SD卡。 打开USB连接。1 2 3 4 5 1:USB已连接电脑(当连接360手机助手)2:作为USB存储设备使用 4:已连接到 USB调试 5:已连接USB
3)连接后可以直接在PC端查看相机拍摄的图片 ?注意:不同的个人电脑操作系统如何操作正常使用U盘。 1)这个主题可以直接使用 2)xp更新windows媒体播放器到11 3)安装wpdmtp。inf司机 4)vista未经证实的 ?注意:在个人电脑业务助理工具如手机,必须打开USB调试。 WLAN提供最远300英尺(100M)的无线网络接入范围。若要使用手机上的WLAN,您必须连接到无线接入点或「热点」。 注意:WLAN信号的可用性与涵盖范围需视数量、基础结构,以及其它信号穿透的对象而定。 开启WLAN并连接到无线网络 1)按下首页>菜单,然后触碰设置 2)在无线和网络下。点击WLAN开关按钮,以开启WLAN。手机会自动扫描可用无线网络。 3)触碰WLAN,进入WLAN设置。接着WLAN网络列表会显示查找到的WLAN网络的网络名称和安全性设置(开 放网络或以WEP、WPA/WPA2加密)。默认启用WLAN高级设置中的网络通知,手机会在查找到有可用的 开放无线网络时在状态栏显示图标。 4)触碰其中一个WLAN网络,以进行连接。当您选取开放网络时,手机会自动连接到该网络。如果选取的 是WEP、WPA/WPA2加密网络,则必须先输入相应的密码,然后再触碰连接。 注意:当手机连接到无线网络后,状态栏会显示
Vector_NTI_中文使用说明书
Vector NTI 7.0 User's Manual 软件包中文翻译者:宋厚辉(浙江大学) 前言(Introduction) 1.程序附带的数据库(Vector NTI database)包括:DNA/RNA、蛋白质、内切酶、寡核苷 酸、凝胶mark。此外程序还提供数据库开发(Database Explorer)功能,用户可以自己修改、添加、拷贝感兴趣的各类数据库。 2.创建新分子(有四种方法) A.用GenBank/GenPept, EMBL/SWISS-PROT and FASTA、ASCII等格式输入DNA 或氨基酸。 B.手工粘帖,然后保存到数据库中 C.从其他分子、接头、载体中剪切、拼接构键 D.从DNA或RNA分子的编码区翻译成蛋白质 3.关于新分子的序列特征图谱:利用GenBank/GenPept, EMBL/SWISS-PROT or FASTA等 格式输入的分子都能显示出序列和结构图,但自己手工粘帖的没有,需要自己编辑 第一章Chapter 1 Tutorial: Display Windows(显示窗口) 目的:创建显示窗口,并对图、序列和文本进行操作 ? 1.登录Vector NTI 安装后首次登录,系统将提示是否允许填充空库,点OK。这样DNA molecules, proteins, enzymes, oligos, and gel markers将组成NTI的数据库。并出现下列两个窗口。 ? 2. 观察出现的Vector NTI 工作窗口和Database Explorer窗口
上面的第一个窗口为工作窗口,由菜单栏和工具条两栏,移动鼠标到工具栏任意选项处,鼠标自动显示每个工具条的功能。 第二个窗口为exlporing——local vector NTI database,显示的是上次打开的DNA/RNA或蛋白分子。 3. Create a Display Window for pBR322 激活exlporing——local vector NTI database窗口中的DNA/RNA Molecules (MAIN) 数据库,找到pBR322分子并双击打开。显示如下窗口:
Winplas中文使用说明书
本程序用来绘制发表质量的质粒图,可广泛应用与论文、教材的质粒插图。其特性包括: ●知道序列或不知序列结构均能绘制质粒图 ●可读入各种流行序列格式文件引入序列信息。 ●自动识别限制位点 ●可构建序列结构,功能包括:从文件插入序列、置换序列、序列编辑、部分序列删 除等。 ●绘图功能强大,功能包括:位点标签说明、任意位置文字插入、生成彩图、线性或 环形序列绘制、可输出到剪贴板、可输出到图像文件。 ●限制酶消化分析报告输出与序列输入报告功能。 1. 不知序列结构绘制质粒图 从菜单或工具栏选择File New,出现一个空白文件,再在菜单或工具栏选择Insert Blank,出现一个对话框,如下: 输入质粒标题与碱基对数,选择为线性或环形序列,单击OK键,便出现质粒图,可继续进行编辑,但因为不知其序列,所以有一些功能无法进行。 2. 从一个GenBank文件建立一个质粒图 选择File New后,在菜单或工具条中选择Insert GenBank File,便打开对话框,由你选择一个指定文件,确定后,便根据此序列生成质粒图,进行进一步编辑。 3. 从一个其它序列格式文件建立质粒图 选择File New后,在菜单或工具条中选择Insert Sequence File,便打开对话框,由你选择一个指定文件,确定后,便根据此序列生成质粒图,进行进一步编辑。支持的序列格式包括:Stanford, NRBF, EMBL, Fasta, GCG, DNAStrider, Fitch, Pearson, Zuker, Olsen, Phylip, ASXII/RAW, PIR, CODA TA, MSF, DNAStar,若是多序列文件,将由你选择哪一条序列进行作图。 4. 标记限制酶切位点 菜单或工具条中中选择Restriction Enzymes命令,用来标记目前序列中发现的限制酶切位点。出现以下窗口:
6-2海德汉中文使用说明书
125 6.4 仿型路径—直角座标 路径功能的概述 直线L 直线 直线终点座标 倒角:CHF 两条直线交点处倒角 倒角边长 圆心CC 刀具不移动 圆心或极心座标 圆C 围绕圆心CC 圆弧移动到圆弧终点 圆弧终点座标,转动方向 圆弧CR 确定半径的圆弧 圆弧终点座标,圆弧半径,转动方向 圆弧CT 和前后型面切线连接的圆弧移动 圆弧终点座标 圆角RND 和前后型面切线连接的圆弧移动 修圆的圆角半径 FK 自由编程 和前一个型面任意连接的直线或者圆弧移动 参阅144页“仿型路径—FK 自由仿型编程”
126 直线L 刀具沿着直线从当前位置移动到直线结束点,该直线的起始点为前一行程序的结束点。 直线结束点的座标 必要时进一步输入: 半径补偿RL/RR/R0 进给率F 辅助功能M NC 程序实例 实际位置归零 您也可用ACTUAL-POSITION-CAPTURE (实际位置归零)键建立直线程序行: 在手动操作模式中,把刀具移动到您要归零的位 置上。 屏幕显示切换到编程和编辑。 选定您要插入L 程序行位置的前一程序行。 按下实际位置归零键:TNC 用当前 的实际位置的座标建立一行程序。, 在MOD 功能中,规定保存在L 程序行中的轴的数量(参阅398页“MOD 功能)。
在两条直线之间插入倒角CHF 倒角功能可以使您切去两条直线交点处的尖角。 ?CHF前后的程序行必须是在同一个平面中的。 ?CHF前后的半径补偿必须相同。 ?内倒角必须足够大,以能容纳在用的刀具。 倒角边长:倒角长度 必要时进一步输入: 进给率F(只在CHF程序行中有 效)。 NC程序行实例 ?您不能用CHF程序行开始仿 型。 倒角只能在加工面中。 尖角被修平,不作为轮廓的一 部分。 CHF程序行中编程的进给率只 对该程序行有效,在CHF程序行以 后,原先编程的进给率恢复有效。 127
pajek 中文使用手册
Pajek 分析和可视化大型网络的程序 参考手册 List of commands with short explanation version 1.16 Vladimir Batagelj and Andrej Mrvar 翻译:先红、一生有我、傻大师、沧海回眸、AndyChang、comp network、遥遥、大头、三叶草 整理:饭团 Ljubljana, October 4, 2006 1996, 2006 V. Batagelj, A. Mrvar. Free for noncommercial use. PdfLaTex version October 1, 2003
Vladimir Batagelj Department of Mathematics, FMF University of Ljubljana, Slovenia http://vlado.fmf.uni-lj.si/ vladimir.batagelj@fmf.uni-lj.si Andrej Mrvar Faculty of Social Sciences University of Ljubljana, Slovenia http://mrvar.fdv.uni-lj.si/ andrej.mrvar@fdv.uni-lj.si
目录 1.Pajek介绍 (1) 2.数据对象 (3) 3 主窗口工具栏 (7) 3.1 File(文件) (7) 3.2 Net(网络) (11) 3.3 Nets(网) (26) 3.4 Operation(操作) (28) 3.5 Partition(分类) (34) 3.6 Partitions(分类) (35) 3.7 Vector(向量) (35) 3.8 Vectors(向量) (36) 3.9 Permutation(排序) (37) 3.10 Cluster(类) (37) 3.11 Hierarchy(层次) (37) 3.12 Options(选项) (38) 3.13 Info(信息) (40) 3.14 Tools(工具) (40) 4 绘图窗口工具 (42) 4.1 主窗口绘图工具 (42) 4.2 Layout(布局) (42) 4.3 Layers(图层) (43) 4.4 GraphOnly(仅图形) (44) 4.5 Previous(退回到前一次操作) (44) 4.6 Redraw(重绘) (44) 4.7 Next(下一步) (44) 4.8 Options(选项) (45) 4.9 Export (导出) (47) 4.10 Spin(旋转) (49) 4.11 Move(移动) (49) 4.12 Info (信息) (49) 5 Exports to EPS/SVG/VRML (50) 5.1 Defaults (默认值) (50) 5.2 Parameters in EPS,SVG and VRML Defaults Window(在EPS/SVG/VRML默认窗口中 的参数) (50) 5.3 Exporting Pictures to EPS/SVG — 在输入文件中定义参数 (52) 6 在Pajek中使用Macros(宏) (57) 6.1 什么是Macro(宏)? (57) 6.2 怎样标明一段宏? (57) 6.3 如何运行宏? (57) 6.4 例子 (57) 6.5 重复最后的命令 (57) 附加信息 (59)
Mapmaker3.0 winQTLCart2.0简单中文教程和举例
内 部使 用 Mapmaker3.0 winQTLCart2.0简单中文教程 和举例 胡磊 (新疆农业大学农学院棉花遗传育种2006级研究生) 本文是笔者自己的试验方法记录里得来的,尽量使用通俗易懂的语言,配合截图,基本上看过的人都可以学会使用。但是由于时间紧促,难免存在疏漏和错误的地方,欢迎指正! 此资料仅供参考,因此引起的后果概不负责,特此声明。 一、前期数据准备 1 数据调查和采样 数据调查:我们把群体中的每个材料种成行,比如3m 一行,在这一行里选取10株做调查,已果枝始节为例,调查10株,并记录在记录本或者电脑里备用。 采 样:在上面说的3m 一行的里取指甲盖大小的叶片,回实验室提NDA ,跑电泳,和读带。 2 数据输入 2.1 大田数据 先把你的田间调查数据输入excel ,以f2代果枝始节调查数据为例。在最后一列算出平均数。注意!我们需要用的就是这个平均数!数据输入的时候,材料编号那里不能出错。 2.2 实验室数据 然后就是我们采回来的叶片跑的电泳记录,这里的叶片都是大田里取来的,全部需要提取DNA 和跑电泳。最后在所跑出来的电泳条带上读取数据,读取数据的方法如下: 如图所示,此图显示的是凝胶上的带型。
内 部使 用 共显性标记:母本带型记为“B ”,父本带型记为“A ”,杂合带型记为“H ”。显性标记记载有两种情况:若母本等位基因为显性,则父本带型记为“A ”,杂种带型记为“D ”;若父本等位基因为显性,则母本带型记为“B ”,杂种带型记为“C ”。无论在显性标记还是共显性标记中,由于某些原因造成的带型不清或数据缺失均用“–”表示。 而我们在读取的时候一般用数字表示,即:1=A 2=B 3=H 4=C 5=D 。在读带后的数据输入也要注意编号,编号一定不能出错。 3 数据处理 3.1 数据核对 将DNA 的编号和大田数据进行对比,如果DNA 里有的编号,在材料编号里没有,就把那一行数据用“-”表示,单元格下移。如果材料编号里有的数据,而DNA 里没有。那就把材料编号和平均数数据一起删除,原则是以DNA 数据为主,同时和大田调查的农艺性状数据一一对应。如图: 3.2 检验数据 调查的数据如果不符合正态分布是不能引用的,数据是否符合正态分布可以用EXCEL
Openbravo中文使用手册
1.1权限管理 定制权限操作演示: 在游览器中输入openbravo后台的登陆用户名和密码,进行操作。 在进行采购管理操作前,先设置下权限,使得你有更大权限进行操作。点击左上角的Openbravo进行权限设定。 在作用这一栏里选择big bazaar admin,在点击OK。这样你拥有最大的权限了。
1.2采购管理 采购管理(Procurement Management) 采购管理是计划下达、采购单生成、采购单执行、到货接收、检验入库、采购发票的收集到采购结算的采购活动的全过程,对采购过程中物流运动的各个环节状态进行严密的跟踪、监督,实现对企业采购活动执行过程的科学管理。 以下是Openbravo采购管理的操作演示。 采购管理—交易操作演示 在采购管理模块下包含有两个子菜单分别为:交易和报表。在交易菜单下的一级目录有:申请、采购申请管理、采购申请、采购订单、物料收货、发票(供应商)、已匹配采购订单、已匹配发票、发货产生的发票、待审收料单 提示:解释下交易和报表这两个菜单的关系,报表菜单的产生是附于交易菜单,报表菜单是可查看交易菜单中的内容。 请购管理—操作演示 是对企业的采购计划进行制定和管理,为企业提供及时准确的采购计划和执行路线。采购计划包括定期采购计划(如周、月度、季度、年度)、非定期采购任务计划(如系统根据销售和生产需求产生的)。通过对多对象多元素的采购计划的编制、分解,将企业的采购需求变为直接的采购任务,系统支持企业以销定购、以销定产、以产定购的多种采购应用模式,支持多种设置灵活的采购单生成流程,采购申请单是分公司根据业务需要,要求总公司进货的单据。采购申请也是采购订单的形成前提。 点击一级菜单“采购管理”,在点击二级菜单“交易”,再点击“交易”下的子菜单“请购管理”,进入“请购管理”页面。 点击左上角上的新建,对请购管理管理表进行编辑,输入:客户、公司、是否有效、业务伙伴、价目表、货币种类、描述信息、等信息后,点击保存 注1:在进行编辑时,可不用手动录入信息,直接下拉选框进行选择。 注2:若在进行编辑时,对于存在的信息还存在一定疑问,在保存时请选择好文档的状态、可保存为草稿的状态。 注3:文档一经删除,将无法恢复,请谨慎操作。 采购订单—操作演示 以采购单为源头,对从供应商确认订单、发货、到货、检验、入库等采购订单流转的各个环节进行准确的跟踪,实现全过程管理。通过流程配置,可进行多种采购流程选择,如订单直接入库,或经过到货质检环节后检验入库等,在整个过程中,可以实现对采购存货的计划状态、订单在途状态、到货待检状态等的监控和管理。采购订单可以直接通过电子商务系统发向对应的供应商,进行在线采购。 点击一级菜单“采购管理”,在点击二级菜单“交易”,再点击“交易”下的子菜单“采购申订单”,进入“采购订单”页面。 点击左上角上的新建,对采购订单进行编辑,输入:业务伙伴、用户/联系人、发票接收地址、价目表、仓库服务点、销售代表、货币、付款规则、打折折扣、付款规则、付款条款、引用的信息、状态的信息后,一切确认无误后,点击保存。 注1:在进行编辑时,可不用手动录入信息,直接下拉选框进行选择。 注2:若在进行编辑时,对于存在的信息还存在一定疑问,在保存时请选择好文档的状态、可保存为草稿的状态。 注3:文档一经删除,将无法恢复,请谨慎操作。 功能按钮说明
Vector NTI7.0中文使用说明书
Vector NTI7.0 User's Manual 软件包中文翻译者:宋厚辉(浙江大学),记住这个伟大的人物吧! 前言(Introduction) 1.程序附带的数据库(V ector NTI database)包括:DNA/RNA、蛋白质、内切酶、寡核苷 酸、凝胶mark。此外程序还提供数据库开发(Database Explorer)功能,用户可以自己修改、添加、拷贝感兴趣的各类数据库。 2.创建新分子(有四种方法) A.用GenBank/GenPept, EMBL/SWISS-PROT and FASTA、ASCII等格式输入DNA 或氨基酸。 B.手工粘帖,然后保存到数据库中 C.从其他分子、接头、载体中剪切、拼接构键 D.从DNA或RNA分子的编码区翻译成蛋白质 3.关于新分子的序列特征图谱:利用GenBank/GenPept, EMBL/SWISS-PROT or FASTA等 格式输入的分子都能显示出序列和结构图,但自己手工粘帖的没有,需要自己编辑 第一章Chapter 1 Tutorial: Display Windows(显示窗口) 目的:创建显示窗口,并对图、序列和文本进行操作 ? 1.登录Vector NTI 安装后首次登录,系统将提示是否允许填充空库,点OK。这样DNA molecules, proteins, enzymes, oligos, and gel markers将组成NTI的数据库。并出现下列两个窗口。 ? 2. 观察出现的Vector NTI 工作窗口和Database Explorer窗口
上面的第一个窗口为工作窗口,由菜单栏和工具条两栏,移动鼠标到工具栏任意选项处,鼠标自动显示每个工具条的功能。 第二个窗口为exlporing——local vector NTI database,显示的是上次打开的DNA/RNA或蛋白分子。 3. Create a Display Window for pBR322 激活exlporing——local vector NTI database窗口中的DNA/RNA Molecules (MAIN) 数据库,找到pBR322分子并双击打开。显示如下窗口:
mapchart使用说明
1.连锁图的绘制 格式标记名称遗传位置颜色(C1、C2、C3等)如不显示标记名称则加o TG580_1.131 0.000c2 b I E36/M51-9p 6.600 TG255_1.117 9.000 E36/M54-2p 18.200 E35/M47-12e 20.200 E35/M47-9p 21.400 TG220_6.100 24.000 TG465_1.90 29.500 TG71_1.58 49.200 TG59_1.52 49.200 E35/M61-13e 56.700 TG273_1.36 56.700 E35/M48-5e 59.000 如让连锁图某个区域,或整段高亮则用命令:segments 区域(起止位置)颜色,如: TG580_1.131 0.000c2 b I E36/M51-9p 6.600 TG255_1.117 9.000 E36/M54-2p 18.200 E35/M47-12e 20.200 E35/M47-9p 21.400 TG220_6.100 24.000 TG465_1.90 29.500 TG71_1.58 49.200 TG59_1.52 49.200 E35/M61-13e 56.700 TG273_1.36 56.700 segments 6.6 49.2 c3
QTL的标注: 格式: Qtls QTL名称外层区域起点内层区域起点内层区域止点外层区域止点,如: TG580_1.131 0.000c2 b I E36/M51-9p 6.600 TG255_1.117 9.000 E36/M54-2p 18.200 E35/M47-12e 20.200 E35/M47-9p 21.400 TG220_6.100 24.000 TG465_1.90 29.500 TG71_1.58 49.200 TG59_1.52 49.200 E35/M61-13e 56.700 TG273_1.36 56.700 segments 6.6 49.2 c3 Qtls pb-4 25.5 30.9 43.3 50.5 BI C2 L3 vg-1 10.0 15.0 35.0 40.0 C3 F4
- 便携式光谱仪 中文使用说明书 正式版
- B-SQUARE码表中文使用说明书
- 绕膜机中文使用说明书
- HDevelop简体中文使用手册
- 使用说明书(中文版) 中英对照
- Wii中文使用说明书
- Niton 中文使用说明书 正式版
- Winplas中文使用说明书
- 中文使用说明书(1)
- DNAStar中文使用说明书
- tree中文使用说明书
- DM中文使用说明书
- Motion 5中文使用手册
- TracePro中文使用手册(上)
- TG39中文使用手册
- 森海塞尔EW中文使用说明书完整版
- 功率计E4418B中文使用说明书
- 中文使用说明书
- 中文使用说明书(1)
- 中文使用说明书(1)报告
- 员工心态管理的方法
- 培养员工积极心态的方法
- 企业领导者应怎样注重员工心态的把握和管理
- 公司员工心态培训
- 把握新员工的四个心理阶段
- 员工心态管理的技巧
- 铝合金水冷缸头低压铸造缺陷分析及优化
- 紫铜结晶器低压铸造缺陷原因分析与对策
- 铸造的种类与优缺点简介[整理]
- 压铸件缩孔缩松的原因及解决办法
- 低压铸造镁合金轮毂裂纹缺陷的改善
- 高压铸造与低压铸造区别
- 生产过程中常见的铸造方法及其优缺点
- 低压铸造铝合金缺陷分析与热处理工艺分析
- 低压铸造铸件缺陷产生原因及防治方法
- 低压铸造优缺点及应用
- 高压铸造与低压铸造区别
- 低压铸造镁合金轮毂裂纹缺陷的改善
- 分析低压铸造法冷隔缺陷的成因及对策
- 低压铸造ZL201A铸件缺陷分析及工艺优化
